Archive된 사유는 다음 중 하나에 해당 됩니다.
- 작성 된지 너무 오랜 시간이 경과 하여, API가 변경 되었을 가능성이 높은 경우
- 주인장의 Data Engineering으로의 변경으로 질문의 답변이 어려워진 경우
- 글의 퀄리티가 좋지 않아 글을 다시 작성 할 필요가 있을 경우
Normalization, PCA
안녕하세요? Justkode 입니다. 이번 시간에는 **Normalization (정규화)**와, 차원 축소를 위한 PCA에 대해 공부 해 보는 시간을 가져 보도록 하겠습니다. 정규화와 차원 축소는 기계 학습에서 중요 한 요소 입니다. 학습에서 직접적인 영향을 주기 때문이죠.
Normalization
저번에 Linear Regression의 Cost Function에 대한 미분을 하게 되면, Input Vector가 나온다는 말을 한적이 있습니다. 다음과 같이 말이죠.
만약 라고 가정 하면, 식을 보면 알다 싶이, 특정 feature의 범위가 0100, 01 이렇게 들쑥 날쑥하게 되면, 똑같은 학습률을 바탕으로, 다른 정도로 학습이 되기 때문에 정규화 과정이 필요 합니다.
Min - Max 정규화
일단 첫 번째로는 Min - Max 정규화가 있습니다. 데이터를 최솟값과 최고값을 이용하여 정규화 하는 방법이며, 이렇게 정규화를 진행 하면 데이터의 범위가 [0, 1] 로 변하게 됩니다.
이를 코드로 구현 하면 다음과 같습니다. numpy.ndarray를 정규화 한다고 가정 하겠습니다.
- Module Import
import numpy as np
import pandas as pd- In
before_n = np.random.normal(0, 10, size=(10,))
before_n- Out: 정규화 전
array([ -7.60552157, -7.4768014 , 3.58856804, -8.64994768,
-5.43849334, 11.49147181, 0.71011417, -1.98272815,
-10.6252785 , -14.26019951])- In
after_n = (before_n - before_n.min()) / (before_n.max() - before_n.min())
after_n # numpy의 Broadcast를 이용하여 간편하게 구현 가능.- Out
array([0.25841732, 0.26341584, 0.69311103, 0.21785972, 0.3425683 ,
1. , 0.58133367, 0.47676406, 0.14115282, 0. ])Z-Score Normalization
두 번째는 Z-Score Normalization 입니다. 이는 입력 데이터의 표준 편차와 평균을 이용하여 구할 수 있습니다. 이를 통해 정규화를 진행 하면, 데이터를 중간으로 모으는 효과를 가질 수 있습니다.
이를 코드로 구현 하면 다음과 같습니다. numpy.ndarray를 정규화 한다고 가정 하겠습니다.
- In
before_n = np.random.normal(5, 10, size=(10,))
before_n- Out: 정규화 전
array([ 3.88259694, -12.78964916, 11.31123123, -1.39481419,
12.67724562, 12.82906507, 6.78477706, -3.9266831 ,
26.89122036, 13.87650172])- In
after_n = (before_n - before_n.mean()) / before_n.std() # mean(): 평균, std(): 표준편차
after_n- Out
array([-0.29603595, -1.87211833, 0.40621733, -0.79492702, 0.53535118,
0.54970317, -0.0216833 , -1.03427294, 1.87904506, 0.64872081])PCA (Principal Component Analysis)
PCA를 한국 말로 나타내면 주성분 분석 입니다. 이는 분포된 데이터들을 통해, 분포의 주성분을 찾아 줌으로써, 가장 큰 분산을 가진 주성분 (분산이 클 수록, 차원을 이동하더라도 정보의 손실이 적음)에 대해 사영 함으로써, 데이터의 차원을 줄이는 방법 중 하나 입니다.
numpy 만으로 구현하기
일단 코드를 먼저 보여 드리겠습니다. np.random.multivariate_normal 를 이용하여, [5, 5]를 평균으로 하고 [[1, 2], [2, 1]] 의 Covariance Matrix를 가지는 50개의 샘플을 만들어 보겠습니다.
- In
import matplotlib.pyplot as plt
array = np.random.multivariate_normal([5, 5], [[1, 2], [2, 1]], 50)
plt.xlim(-5, 10)
plt.ylim(-5, 10)
plt.scatter(array.T[0], array.T[1])
plt.show()- Out
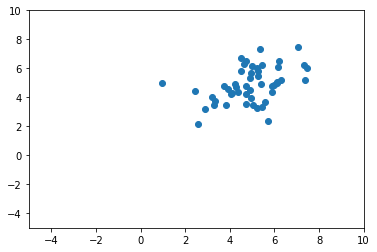
초기 데이터
그 다음, 샘플들을 Broadcasting 을 이용하여, 데이터를 원점으로 모아 줍니다.
- In
array -= np.array([array.T[0].mean(), array.T[1].mean()])
plt.xlim(-5, 5)
plt.ylim(-5, 5)
plt.scatter(array.T[0], array.T[1])
plt.show()- Out
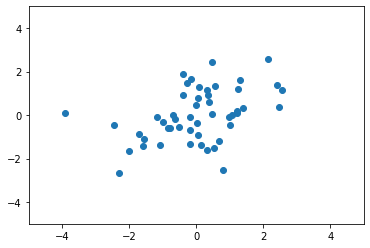
중앙으로 모아줌
그 다음, 생성된 데이터에 대한 Covariance Matrix를 구하고, 이에 대한 고유 벡터와 고유값을 구해 줍니다. 여기서 고유값이 높다는 것은, 이에 대응하는 **고유 벡터 (혹은 주성분)**에 각 Input Vector를 사영 할때, 얼마나 분산이 높은지? 그리하여, 어떻게 정보의 손실없이 사영할 수 있는 지를 나타냅니다. 다시 한번 정리 하자면, 고윳값이 높을 수록, 이에 대응하는 고유벡터를 주성분으로 삼았을 때, 정보의 손실이 작다는 것을 의미 합니다.
- In
cov = np.cov(array.T)
eigen_val, eigen_vec = np.linalg.eig(cov)
plt.scatter(array.T[0], array.T[1])
plt.quiver([0, 0], [0, 0], eigen_vec[:, 0], eigen_vec[:, 1], color=['r', 'b'], scale=3)
print("eigenvalue: ", eigen_val)
print("eigenvector: ", eigen_vec)- Out
eigenvalue: [2.24427897 0.89684849]
eigenvector: [[ 0.76269005 -0.64676417]
[ 0.64676417 0.76269005]]
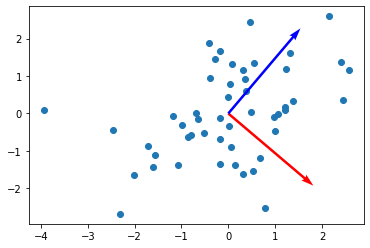
Eigenvector == 주성분
마지막으로, 해당 주성분에 벡터들을 사영을 해주면 완성이 됩니다. argmax() 메서드를 이용하여, 주 성분으로 뽑을 고유 벡터를 선택합니다.
- In
index = eigen_val.argmax()
v = eigen_vec[index, :] # norm: 1
result = array.dot(v) * v.reshape(-1, 1)
plt.scatter(result[0], result[1], color='green')
plt.scatter(array.T[0], array.T[1])
plt.quiver(0, 0, v[0], v[1], color='black', scale=4)
plt.xlim(-4, 4)
plt.ylim(-4, 4)
plt.show()- Out
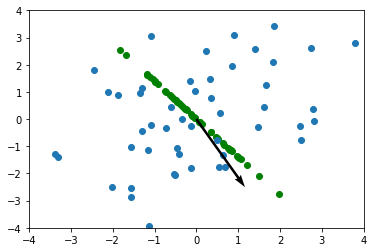
사영 한 모습
sklearn으로 더 쉽게 구현하기
이는 Scikit-Learn으로 더 쉽게 구현 할 수 있습니다. sklearn.decomposition 내의 PCA를 이용합니다. n_components를 이용 하여, 몇 차원으로 줄이고 싶은지 입력 할 수 있습니다.
- In
from sklearn.decomposition import PCA
pca = PCA(n_components=1) # 차원 수 1개
pca.fit(array)
print('고유 값 :', pca.explained_variance_) # 고윳값
print('고유 벡터 :', pca.components_.T) # 고유 벡터
print('사영 후:', pca.transform(array))- Out
고유 값 : [2.6328486]
고유 벡터 : [[0.81183442]
[0.58388772]]
사영 후: [[ 1.1017955 ]
[ 1.90965349]
[-1.26196986]
[ 0.47308556]
...
[ 0.83702101]
[-1.95731602]]마치며
이번 시간에는 Normalization, PCA에 다뤄 보았습니다. 다음 시간에는 딥러닝의 제일 기본이 되는, DNN에 대해 공부해 보는 시간을 가져 보도록 하겠습니다.